Pharmaunternehmen verfügen über eine Unmenge an internen wie externen Daten. Doch Daten allein stellen noch kein Wissen dar und das Verknüpfen und Aktualisieren von unterschiedlichen Datenquellen kann schnell zur Sisyphusarbeit ausarten. Das liegt zum einen an der schieren Menge an Informationen. Zum anderen sind die Daten oft heterogen und liegen in unterschiedlichen Formaten vor.
Ohne technische Unterstützung ist es für Pharmaforscher heute schlichtweg unmöglich, mit dem Wissenswachstum Schritt zu halten. Zumal es in der Medizin verstärkt darum geht, interdisziplinäre Ansätze voranzutreiben, um Zusammenhänge zwischen verschiedenen Krankheiten und Fachgebieten aufzudecken. Viele Pharmaunternehmen sitzen zudem auf Legacy-Daten, die in Datensilos über Jahrzehnte hinweg ungenutzt bleiben.
Künstliche Intelligenz, Machine Learning und Natural Language Processing (NLP) haben in den letzten Jahren die Arbeitsweise in der Pharmaforschung grundlegend verändert. Doch auch diese Technologien brauchen zunächst ein Daten-Framework, um die vielfältigen Informationen zu einem Gesamtbild zusammenzufügen und aus reinen Daten Wissen zu generieren.
Knowledge Graph für mehr Kontext
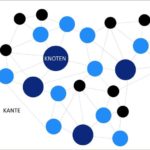
Knowledge Graphen bieten genau ein solches Datenmodell. Anders als relationale Datenbanken sind sie darauf ausgelegt, Daten in ihrer ganzen Komplexität und Reichhaltigkeit abzubilden. Aus diesem Grund speichert der Knowledge Graph nicht nur einzelne Daten (Knoten), sondern auch die Beziehungen zwischen ihnen (Kanten) ab. Jedem Knoten und jeder Kante lassen sich dabei beschreibende Eigenschaften zuweisen. Das intuitive Knoten-Kanten-Prinzip macht es sehr einfach, neue Daten hinzuzufügen und selbst komplexe Zusammenhänge anschaulich zu visualisieren. Der Knowledge Graph ist unbegrenzt skalierbar, wächst mit jeder Abfrage und entwickelt sich so zum zentralen Wissenshub für Forscher innerhalb eines Unternehmens.
Vor allem schafft der Knowledge Graph den Kontext, den Pharmaforschern benötigen, um schneller und zielgerichteter Wechselwirkung von medizinischen Wirkstoffen und therapeutischen Angriffspunkten (Targets) zu erforschen. Als Data Scientist folgen sie den Verknüpfungen über mehrere Knoten hinweg, tauchen in Datengruppen ein, bewegen sich frei in alle Richtungen vor und zurück und haben dabei stets Zugriff auf die neusten biomedizinischen Daten.
Novartis biologisches Wissen als Graph
Der Schweizer Pharmariese Novartis entwickelte mit Hilfe der Graphdatenbank Neo4j eine solche zentrale Wissensplattform. In ihrem Knowledge Graphen sind unterschiedliche Datenquellen miteinander verknüpft, um das biologische Gesamtwissen des Unternehmens Mitarbeitern zur Verfügung zu stellen. Neben der rund einen Milliarde an Legacy-Daten aus der Forschung gehören dazu auch Terabytes an phänotypischen Daten, die Novartis über ein automatisiertes Verfahren erhebt. Die internen Daten werden dabei kontinuierlich in den Kontext der weltweiten medizinischen Forschung gerückt.
Die Forscher definierten zunächst ein Datenmodell mit 15 Knoten-Kategorien sowie 90 möglichen Datenbeziehungen (Kanten) und machten sich anhand dieses Bauplans an die Migration der Legacy-Daten sowie der kontinuierlich wachsenden Menge an Bilddaten. Über Text Mining werden zudem relevante Information aus der PubMed extrahiert und im Graphmodell abgelegt. Die Literaturdatenbank des National Institutes of Health (NIH) bietet Forschern Zugang zu etwa 25 Millionen Texten aus über 5600 wissenschaftlichen Zeitschriften. Zu Beginn umfasste der Knowledge Graph von Novartis eine halbe Milliarde Beziehungen – eine Zahl, die sich jedoch sehr schnell verdreifachte.
Forschen im Dreieck aus Target, Krankheit und Wirkstoff
Mit dem Knowledge Graph von Neo4j ist Novartis in der Lage, in allen Datenquellen flexibel zu navigieren. Graph-Algorithmen vereinfachen und beschleunigen die Abfragen im Knoten-Dreieck aus Target, Krankheit und Wirkstoff zusätzlich. So lässt sich beispielsweise untersuchen, wie stark die Verbindungen innerhalb eines Knoten-Dreiecks und damit die Erfolgsaussichten eines möglichen Wirkstoffs sind. Diese Analyse funktioniert auch in umgekehrter Richtung: Indem die Forscher die Verbindungen nach ihrem Stärkegrad filtern, stoßen sie neben bekannten Korrelationen auf neue Zusammenhänge, die ihre Forschungsarbeit in unerforschte Richtungen lenken.
Die riesige Menge an biomedizinischen Daten lässt sich damit erstmals in ihrer gesamten Komplexität für die Medikamentenentwicklung nutzen. Die zentrale Ansicht liefert schnell und zuverlässig Antworten auf elementare Forschungsfragen. Welcher Wirkstoff ähnelt Wirkstoff A, der mit der Krankheit X verbunden ist? Wie stark ist dort die Verbindung zwischen Target und Wirkstoff? Gibt es bereits vergleichbare Untersuchungen zum Wirkstoff?
Wissenshub für die Diabetesforschung
Mit ähnlichen Fragen beschäftigt sich auch das Deutsche Zentrum für Diabetesforschung (DZD). Welcher Diabetes-Typ lässt sich auf welche Gene zurückführen? Und wie wirken sich externe Faktoren auf die Krankheit aus? In einem Knowledge Graphen werden Metadaten aus klinischen Studien mit disziplinübergreifenden Forschungsdaten aus öffentlichen Quellen verknüpft. Die humanen Daten werden dabei um hochstandardisierte Daten aus Tiermodellen (z. B. Mäuse) ergänzt, um Rückschlüsse von einem Modellorganismus auf den Menschen ziehen und Ähnlichkeiten bei einzelnen Genen und Stoffwechselvorgängen untersuchen zu können.
Der Knowledge Graph liegt als Layer über den bestehenden Datenbanken des DZDs und wird standortübergreifend von Healthcare und Medical Professionals genutzt. Eine zentrale Rolle übernimmt die Graph Data Science Library von Neo4j. Mit Hilfe der integrierten Algorithmen können die Wissenschaftler das Datenset unterteilen und die verschiedenen Subtypen des Typ-2-Diabetes besser erforschen. So erkennt der Community-Detection Algorithmus zum Beispiel Patienten-Cluster und gibt Aufschluss über entsprechende Charakteristiken (z. B. Größe, Medikation). Auch hier ist das langfristige Ziel, neue Erkenntnisse für die Prävention und Therapie zu gewinnen (Stichwort: Precision Medicine).