Die Leistungsfähigkeit von Chemie-Software für den PC steigt parallel zur Entwicklung der laufend verbesserten Prozessoren. Ein Beispiel ist das PC-Chemieprogramm Alchemy 2000.
Dr. Thomas Beneke, Dr. Wolfgang Schwippert
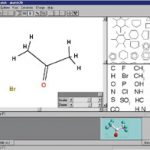
Alchemy übernimmt die grafische Umsetzung chemischer Strukturen in 2D und 3D, die Anordnung der Atome im Molekül gemäß der energetisch optimalen Geometrie und die Berechnung intramolekularer Ladungs- und Energieverteilungen, Bindungswinkel, Bindungslängen, Torsionswinkel o. ä. Die erzeugten Grafiken und die errechneten Parameter lassen sich in verschiedenen Formaten als Ergebnisse eines Einzelprojektes oder einer Stapelverarbeitung für den Routinebetrieb ausgeben.
Tools in jeder Form
Nach dem Programmaufruf zeigen sich die obligatorischen Windows-Bedienelemente wie Menüzeile, Schaltflächenleiste, Arbeitsfläche und Statuszeile. Die automatisch eingeblendete Arbeitsfläche dient der Erzeugung einfacher wie komplexer Molekülansichten in 3D. Sie wird von einer individuell konfigurierbaren vertikalen Werkzeugleiste mit chemiespezifischen Funktionen und einem Hilfsdialog flankiert. Wird hier das Register Elemente angeklickt, öffnet sich eine Werkzeugpalette und erlaubt die Auswahl chemischer Elemente und Bindungskonfigurationen, im Falle des Kohlenstoffs beispielsweise sp3-, sp2- oder sp-Hybridisierungen. Wird das alternative Register Protein aktiviert, erscheint eine Palette mit Aminosäuren gemeinsam mit Schaltfeldern zur Festlegung der räumlichen Strukturierung und der Darstellungsweise von Proteinen. Eine weitere Schaltfläche führt in ein Spreadsheet, das die Aminosäuresequenz der Proteinketten anzeigt, also die Primärstruktur. Hier lassen sich Aminosäuren gezielt auswählen. Auf diese Weise kann beispielsweise in einem mit Strichen räumlich dargestellten Protein die Tertiärstruktur einer bestimmten Aminosäuresequenz in Form von Kalotten gezeigt werden. Damit ist das Angebot an speziellen Hilfsmitteln aber lange nicht erschöpft, denn über die Menüleiste lassen sich auch Dialogfenster aufrufen, in denen per Schiebeschalter u. a. konstante Rotationsgeschwindigkeiten eines Moleküls auf dem Monitor festgelegt werden, so daß sich Animationseffekte ergeben. Andere Dialoge erlauben die Festlegung beliebiger Skalierungs- und Zoomfaktoren.
Eine Reihe von Funktionen für Rotation und Translation werden sehr komfortabel über die sogenannte Kontrollbox bedient. Die Organisation dieser Box erinnert an die Fadenkreuze der komplizierten Zieleinrichtungen einschlägiger Flugsimulationsspiele. In Alchemy 2000 lassen sich damit per Mausklick an Schaltfelder und Skalen alle wichtigen grafischen Anzeigeoperationen für ein Molekül im Raum festlegen, also zum Beispiel Drehungen um die Raumachsen, Zoom, Stereoansicht oder Bewegungen in den Raumrichtungen. Mit einigen traditionellen Schaltflächen der vertikalen Werkzeugleiste werden dagegen einzelne Atome oder funktionelle Gruppen innerhalb molekularer Strukturen bzw. Molekülfragmenten ausgewählt. Im Falle von Proteinen darf noch eine spezifische Markierung des Backbone vorgenommen werden, wobei für den eigentlichen Aufbau und das Editieren des Proteins noch eine weitere spezielle Proteineditorleiste zur Verfügung steht, die u. a. die verwendeten Darstellungsmodi symbolisch anzeigt. Sie blendet sich automatisch ein, sobald ein Protein auf den Monitor zitiert wird. Zu den anwenderfreundlichen Werkzeugen gehört auch noch die rechte Maustaste, mit der sich je nach geöffneter Arbeitsfläche verschiedene Operationen durchführen oder Dialoge und Menüs öffnen lassen.
Um insbesondere die Effekte geringfügiger Manipulationen besser vergleichen zu können, empfiehlt es sich, eine chemische Struktur in mehreren Fenstern gleichzeitig sichtbar zu machen. Werden nun Änderungen am Molekül vorgenommen, erfolgen diese zunächst einmal nur in dem aktiv geschalteten Fenster. Dies erleichtert den Vergleich nahezu gleichartiger Moleküle oder unterschiedlicher Molekülzustände.
2D – 3D – Stereo
Die Darstellung dreidimensionaler Molekularstrukturen ist ein wichtiges Einsatzgebiet für Chemiesoftware. Alchemy 2000 bietet hierzu alle bei Chemikern allgemein akzeptierten Darstellungsformen, wobei grafische Extras wie Schattierungen, Mischdarstellungen und freie Farbwahl für die verwendeten Atome selbstverständlich sind. Besonders hervorzuheben sind kombinierte Anzeigemöglichkeiten. So läßt sich z. B. ein Proteinbackbone als Band und die charakteristischen Aminosäurereste als Röhrchen oder Stäbchen visualisieren. In diese Bilder läßt sich eine Reihe symbolischer und numerischer Informationen einblenden. So dürfen zusätzliche Parameter wie Chiralität, Ladungen, Hybridisierungstypen, Elementnummern oder Elementsymbole plaziert und den entsprechenden Strukturen zugeordnet werden. Was das optische Erscheinungsbild der wichtigsten Formelattribute angeht, lassen sich die relevanten Größen voreinstellen. Dazu gehören Schattierungstiefen, Radien von Kugeln oder Kalotten, die Breite von Bindungen und einiges mehr.
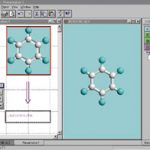
Viel häufiger als Grafiken in 3D werden planare Abbildungen von Strukturformeln benötigt, z. B. für Publikationen. Alchemy 2000 hält hierfür eine spezielle 2D-Arbeitsfläche bereit. Sie besitzt zwar im Prinzip den gleichen Aufbau wie der sogenannte 3D- Builder, unterscheidet sich aber in allen Bedienungselementen wie Menüs und Schaltflächen von diesem. Auch die vertikale Werkzeugleiste zeichnet sich durch völlig andere Funktionen aus. Sie liefert hier insgesamt 24 Schalter, mit denen sich per Knopfdruck alle entscheidenden Manipulationen für das Moleküldesign innerhalb der Zeichenebene vornehmen lassen. So dürfen beispielsweise Bindungstypen eingefügt bzw. entfernt, Atome wegradiert oder Texte integriert bzw. gelöscht werden. Auch in diesem Modus enthält die Monitorfläche auf der rechten Seite eine Hilfspalette, diesmal bestehend aus zwei Bereichen, die hier Molekülvorlagen und -fragmente bereitstellen. Per Drag & Drop lassen sich aus ihnen Formelbestandteile auf die Zeichenfläche kopieren, um sie dort zu überarbeiten und zu neuen Strukturen zusammenzufügen. Die vorgefertigten Zeichenelemente werden einer Datenbank entnommen, die beliebig erweitert werden darf. Auch weitere Fragmente darf der Programmanwender der vorhandenen Bibliothek hinzufügen. Gleiches gilt übrigens für die oben erwähnten Hybridisierungszustände. Die letztlich erzeugten Strukturformeln lassen sich von der planaren Anordnung in die Raumstruktur konvertieren, wobei das Programm automatisch zur 3D-Arbeitsfläche wechselt.
Berechnungen für den Export
Alchemy 2000 ist sehr kommunikationsfreudig. So lassen sich Daten in 18 Formaten importieren, und es stehen 25 Dateiformaten für den Export zur Auswahl. Außerdem lassen sich Daten und Strukturen als Windows-Metadateien, als Bitmaps oder in Form anderer Grafikformate auch an fachfremde Software weitergeben. Hinzu kommt, daß numerische Daten in eine Excel-Tabelle einlesbar sind, womit neben Excel selbst auch die Excel-kompatiblen Anwenderprogramme bedient werden können und damit für weitergehende Analysen zur Verfügung stehen. Darüber hinaus läßt sich selbstverständlich auch der Weg über die Zwischenablage wählen, um Datenmaterial in andere Programme zu überführen.
Fähigkeiten zur mathematischenModellierung
Neben der Bestimmung von Bindungslängen und -winkeln berechnet Alchemy zur Beschreibung der molekularen Eigenschaften bis zu 26 Parameter, wobei insbesondere die charakteristischen Energieanteile und -verteilungen von Interesse sind. Van der Waals, Dipolinteraktionen, Schwingungen mögen in diesem Zusammenhang als stichwortartige Hinweise ausreichen. Aus der programminternen Ergebnistabelle dürfen errechnete Daten anschließend in eine Excel-Tabelle exportiert werden, um sie dort weiter auszuarbeiten und gegebenenfalls zu verrechnen. Etwas mehr Mathematik erfordert es, die Gesamtenergie in einem Molekül zu errechnen und dabei zu minimieren, womit gleichzeitig die stabilste Anordnung der einzelnen Atome und Bindungen im Raum gefunden ist. Alchemy 2000 bietet hierzu molekular mechanische und semi-empirische Verfahren an.
Semi-empirische Berechnungsmethoden sind durch die Integration des Programmpakets MOPAC in Alchemy 2000 zugänglich. Sie erlauben die Berechnung von Moleküleigenschaften und Geometrien. Wer molekulare Strukturen gleich serienweise anhand von errechneten Parametern charakterisieren möchte, richtet sich zuerst eine datenbankähnlich strukturierte Liste ein und arbeitet anschließend im sogenannten Batchmode. Die seriell erarbeiteten Daten lassen sich wiederum tabellarisch anzeigen und exportieren, oder aber die errechneten einzelnen Parameter werden einzeln oder gemeinsam in einer Grafik visualisiert.
Weitere Informationen cav-206
Unsere Webinar-Empfehlung
Die Websession „Wasserstoff in der Chemie – Anlagen, Komponenten, Dienstleistungen“ (hier als Webcast abrufbar) zeigt technische Lösungen auf, die die Herstellung und Handhabung von Wasserstoff in der chemischen Industrie sicher machen und wirtschaftlich gestalten.
Ob effizienter…
Teilen: